中科院微生物所在CRISPR适应机制方面取得系列进展
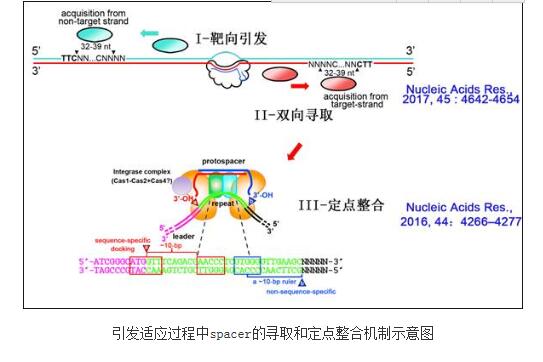
CRISPRs-Cas系统是广泛存在于细菌和古菌中的适应性核酸免疫系统。该系统具有丰富多样的功能组分和核酸处理机制,为人类提供了迄今最高效的基因组编辑技术(如CRISPR-Cas9系统)和基因检测技术(如CRISPR-C2c2/Cas13a系统),同时也为理解生命的进化与适应机制提供了前沿窗口。中国科学院微生物研究所微生物资源前期开发国家重点实验室向华研究组是我国较早从事CRISPR-Cas系统基础研究的团队,面对国际上长期缺乏CRISPR从纯病毒高效获取spacer的适应系统的困境,于2014年在古菌中建立了首个(所有系统中第二个)CRISPR-Cas系统对纯病毒的高效适应体系,揭示了嗜盐古菌I-B型CRISPR高效适应需要“引发”的本质,首次提出了引发适应可能是CRISPR系统在自然界对病毒发生高效适应的主要模式这一重要论断;并进一步发现除高效性外,引发适应还通过巧妙的PAM验证实现了严格的异己区分和高效的防病毒逃逸机制,回答了困扰科学家多年的CRISPR适应过程的高效性及异己区分难题(Nucleic Acids Res., 2014,42:2483–2492;Nucleic Acids Res., 2014,42:7226–7235)。相关工作作为CRISPR适应领域的重要发现已被Nature, Cell, Science, PNAS 等引用80余次。最近,向华研究组通过对该CRISPR系统的人工改建,又在CRISPR适应过程中spacer的长度决定和定点整合的位点识别机制方面连续取得了新的进展(Nucleic Acids Res., 2016,44:4266–4277;Nucleic Acids Res., 2017,45 : 4642-4654)。
CRISPR适应过程中,spacer整合反应往往发生于CRISPR结构的leader端且与其紧邻的repeat将发生精确复制。长期以来,国际上普遍认为整合复合物先识别repeat的一端,再通过严格的分子尺机制识别另一端,从而确定新复制repeat的尺寸和序列。但有意思的是,多个研究团队倾向于先识别序列保守的“repeat近leader”端,而另外有实验室则认为是先识别“repeat远leader”端。向华研究组巧妙设计了一个引发-整合相分离的CRISPR高效适应系统,通过系统的扫描突变鉴定了repeat内部两个关键的整合识别元件。其中,元件1(AACCC)严格位于“近leader”端整合位点的下游10 bp处,而“远leader”端整合反应严格地发生于元件2(GTGGG)下游约10 bp处。上述两元件为repeat识别所必需,且其间距的增减可在一定范围内相应增减repeat的固有尺寸,这说明在spacer整合过程中并不存在repeat长度的分子尺机制,而是整合复合物先识别近leader的repeat的内部关键元件,并通过10-bp左右的分子尺识别两端的整合反应位点(Nucleic Acids Res., 2016,44:4266–4277)。有意思的是,这一重要发现发表不久,很快得到了国际上其他团队在大肠杆菌中实验数据的支持,说明了该机制在不同CRISPR系统中具有普遍性。
关于spacer长度决定机制,2016年国际上两家重要实验室几乎同时解析了大肠杆菌spacer获取机器(Cas1-Cas2复合物)在底物结合状态下的晶体结构,他们发现该复合物的结构性限制提供了一个固定的分子尺,并界定了spacer的长度。但令人费解的是,在其它大多数的CRISPR系统中,spacer长度并非固定不变,而是具有一定的尺寸多态性。向华研究组进一步设计了一个CRISPR单一引发的适应系统,利用高通量测序技术,分析了近4万个新spacer的获取过程,发现与自然界已有的数据一样,这些spacer的尺寸并非固定不变,而是在一定范围内呈现正态分布。有意思的是,他们通过生物信息学分析检测到在spacer倒数第三个碱基位点上的胞嘧啶(C)偏好性,对相应位点的突变则可改变获取spacer的尺寸。该高通量数据结合分子遗传学实验分析,首次发现spacer获取机器不仅识别protospacer 5’一侧的PAM序列,而且识别protospacer 3’端的部分序列,这一序列特异性识别可对获取机器的分子尺机制进行微调,从而导致了spacer尺寸的多态性。该spacer获取的大数据分析工作,还观测到了适应机器在病毒模板上的滑脱(slip)和在整合过程中的翻转(flip)现象,并进一步证明了引发适应过程中双向寻取spacer的滑动(sliding)假说(Nucleic Acids Res., 2017,45 : 4642-4654)。
上述最新进展为系统理解CRISPR引发适应过程(靶向引发,双向寻取,定点整合)的高效性与特异性提供了新的依据(图1),也为未来开发基于CRISPR精妙的适应过程的分子生物学技术奠定了基础。向华团队的李明和龚路遥是Nucleic Acids Res., 2017,45 : 4642-4654的并列第一作者;王锐和李明是Nucleic Acids Res., 2016,44: 4266–4277的并列第一作者;向华是上述论文的唯一通讯作者。相关研究得到了国家自然科学基金(面上项目)的资助。

 打印本文
打印本文 关闭窗口
关闭窗口